Variabilidade Genética de Bovinos das Raças Guzerá e Senepol por Marcadores Microssatélites
DOI:
https://doi.org/10.20873/jbb.uft.cemaf.v4n3.silvafilhoPalavras-chave:
Gado, STRs, diversidade, conservação animalResumo
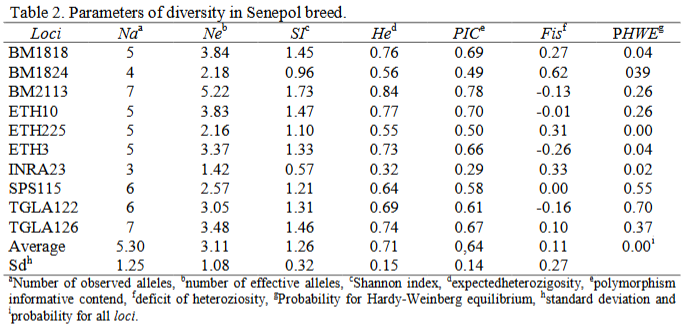
A bovinocultura desempenha função importante na economia Brasileira e as raças Guzerá e Senepol são produtoras de carne. Objetivou-se analisar as variabilidades genéticas das raças Guzerá e Senepol através dos marcadores microssatélites. Os animais foram coletados e genotipados para dez locimicrossatélites em um sequenciador automático de DNA e analisado estatisticamente. Um total de 53 alelos foi observado com número médio de 3,36 para Guzerá e 3,11 para a raça Senepol. Os índices de Shannon foram 1,36 para Guzerá e 1,26 para raça Senepol. As heteroziosidades esperadas foram 0,71 e os valores de PIC foram 0,64 em ambas as raças. Os valores de FISforam 0,01 e 0,11 para Guzerá e Senepol, espectivamente e as proporções de Hardy-Weinberg foram não ignificativas em Guzerá (P>0,05) e significativas em Senepol (P<0,05). Os poderes de discriminação combinados foram 0,99 em ambas as raças e os poderes de exclusões combinados (PE1) foram 0,99 em ambas as raças e os poderes de exclusão combinados (PE2) foram 0,96 e 0,95 em Guzerá e Senepol, respectivamente. Existe variabilidade genética em ambas as raças, mas existem evidências de endocruzamentos por consequência da deriva genética e seria necessário avaliar um maior número de locimicrossatélites para aumentar a eficiência no poder de exclusão (PE2)
Referências
ARIF, I.A.;KHAN, H.A.;SHOBRAK, M.;HOMAIDAN, A.A.;SADOON, M.A.;FARHAN, A.H. Measuring the genetic diversity of Arabian Oryx using microsatellite markers: implication for captive breeding. Genes & Genetic Systems, v.85, p.141-145, 2010.
BOTSTEIN, D.; WHITE, R.L.; SKOLNICK, M.; DAVIS, R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, v.32, p.314- 331, 1980.
CARNEIRO, T.X.; GONÇALVES, E.C.; SCHNEIDER, M.P.C.; SILVA, A. Diversidade genética e eficiência de DNA microssatélites para o controle genealógico da raça Nelore. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v.59, p.1257-1262, 2007.
CERVINI, M.; SILVA, H.F.; MORTARI, N.; MATHEUCCI, J.R.E. Geneticvariabilityof 10 microsatellitemarkers in thecharacterizationofBrazilianNellorecattle (Bosindicus). Genetics and Molecular Biology, v.29, p.486-490, 2006.
CHARLESWORTH, B.; SNIEGOWSKI, P.; STEPHAN, W. The evolutionary dynamics of repetitive DNA in eukaryotes. Nature, v.371, p.215- 220, 1994.
DALVIT, C.; DE MARCHI, M.; DAL ZOTTO, R.; ZANETTI, E.;MEUWISSEN, T.; CASSANDRO, M. Genetic characterization of the Burlina cattle breed using microsatellites markers. Journal of Animal Breeding and Genetics, v.125, p.137–144, 2008.
EDWARDS, A.;CIVITELLO, A.;HAMMOND, H.A.;CASKEY, C.T. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats. American Journal of Human Genetics, v.49, p.746-756, 1991.
EDWARDS, A.; HAMMOND, H.A.; JIM, L. et al.Genetic variation at five trimeric and tetrameric tandem repeat loci in four human population groups. Genomics, v.12, p.241-253, 1992.
EGITO, A.A.; PAIVA, S.R.; ALBUQUERQUE, M.D.S.M.; MARIANTE, A.S.; ALMEIDA, L.D.; CASTRO, S.R.; GRATTAPAGLIA, D.Microsatellite based genetic diversity and relationships among ten Creole and commercial cattle breeds raised in Brazil. BMC Genetics, v.8, p.1-14, 2007.
FATIMA, S.;BHONG, C.D.;RANK, D.N.;JOSHI, C.G. Genetic variability and bottleneck studies in Zalawadi, Gohilwadi and Surti goat breeds of Gujarat (India) using microsatellites. Small Ruminant Research, v.77, p.58-64, 2008.
FISHER, R. Standard calculations for evaluating a blood group system. Heredity, v. 5, p. 95-102, 1951. IBGE, Instituto Brasileiro de Geografia e Estatística. Produção da Pecuária Municipal, Rio de Janeiro, RJ, Brasil, v.39, 2011.63p.
JAMIESON, A.; TAYLOR, S.C. Comparisons of three probability formula for parentage exclusion. Animal Genetics, v.28, p.397-400, 1997.
KANTANEN, J., OLSAKER, I., HOLM, L.E.; LIEN, S.; VILKKI, J.; BRUSGAARD, K.; EYTHORSDOTTIR, E.; DANELL, B.; ADALSTEINSSON, S. Genetic diversity and population structure of 20 north European cattle breeds. The Journal of Heredity, v.91, p.446-457, 2000.
KARTHICKEYAN, S.M.K.; SIVASELVAM, S.N.; SELVAM, R.; THANGARAJU, P. Microsatellite analysis of Kangayam cattle (Bosindicus) of Tamilnadu.Indian Journal of Science and Technology, v.2, p.38-40, 2009.
KLOOSTERMAN, A.D.; BUDOWLE, B.; DASELAAR, P. PCR-amplification and detection of the human D1S80 VNTR locus, Amplification conditions, population genetics and application in forensic analysis. International Journal of Legal Medicine, v.7, p.257-264, 1993.
NEY, M.; ROYCHOUDHURY, A.K. Sampling variances of heterozygosity and genetic distance. Genetics, v.76, p.379-390, 1974.
PAETKAU, D.; CALVERT, W.; STIRLING, I.; STROBECK, C. Microsatellite analysis of population structure in Canadian polar bears. Molecular Ecology, v.4, p.347-354, 1995.
RAYMOND, M.; ROUSSET, F. GENEPOP (Version 1.2): Population Genetics Software for Exact Tests and Ecumenicism. Journal of Heredity, v.86, p.248-249, 1995.
REHMAN, M.S.; KHAN, M.S. Genetic diversity of hariana and hissar cattle from pakistan using microsatellite analysis. Pakistan Veterinary Journal, 29, 67-71, 2009.
ROY, M.S., GEFFEN, E., SMITH, D.; OSTRANDER, E.A.; WAYNE, R.K.Patterns of differentiation and hybridization in North American wolflikecanids, revealed by analysis of microsatellite loci.Molecular Biology and Evolution, v.11, p.553-570, 1994.
RUBINSZTEIN, D.C. Trinucleotide expansion mutations cause diseases which do not conform to classical Mendelian expectations. In: Goldstein, D.B.;Schlötterer, C. (eds) Microsatellites: evolution and applications. Oxford University Press, 1999, pp 80–97.
SAMBROOK, J.; FRITSCH, E.F.; MANIATIS, T. Molecular Cloning. In A Laboratory Manual. Cold Spring Harbor Laboratory Press. New York, v. 1, 2 and 3. 1989.
SERRANO, G.M.; EGITO, A.A.; MCMANUS, C.; MARIANTE, A.S. Genetic diversity and population structure of Brazilian native bovine breeds. PesquisaAgropecuáriaBrasileira, v.39, p.543-549, 2004.
SHEKAR, M.C.; KUMARA, J.U.; KARTHICKEYAN, S.M.K.; MUTHEZHILAN, R. Assessment of with-in breed diversity in Hallikar cattle (Bosindicus) through microsatellite markers. IndianJournalof Science and Technology, v.4, p.895- 898, 2011.
SILVA FILHO, E.; SCHNEIDER, M.P.C.; SILVA, A. Variabilidade Genética de Cavalos baseada em DNA Microssatélites. RevistaTrópica: CiênciasAgrárias eBiológicas, v.1, p.76-87, 2007.
STEVANOVIC, J.; STANIMIROVIC, Z.; DIMITRIJEVIC, V.; MALETIC, M. Evaluation of 11 microsatellite loci for their use in paternity testing in Yugoslav Pied cattle (YU Simmental cattle).Czech Journal of Animal Science, v.55, p.221–226, 2010.
WEIR, B.S.; COCKERHAM, C.C. Estimating F-Statistics for the Analysis of Population Structure. Evolution, v.38, p.1358-1370, 1984.
WEISSENBACH, J., GYAPAY, G., DIB, C.; VIGNAL, A.; MORISSETTE, J.; MILLASSEAU, P.; VAYSSEIX, G.; LATHROP, M. A second-generation linkage map of the human genome. Nature, v.359, p.794–801, 1992.
YEH, F.; YANG, C.; BOYLE, T. POPGENE version 1.32 Microsoft window-based freeware for Population Genetic Analysis. University of Alberta. Edmonton, AB. Canada. 1999.
Publicado
Como Citar
Edição
Seção
Licença
Copyright (c) 2024 - Journal of Biotechnology and Biodiversity

Este obra está licenciado com uma Licença Creative Commons Atribuição 4.0 Internacional.
Autores que publicam nesta revista concordam com os seguintes termos:
Autores mantêm os direitos autorais e concedem à revista o direito de primeira publicação, com o trabalho simultaneamente licenciado sob a Licença Creative Commons Attribution (CC BY 4.0 no link http://creativecommons.org/licenses/by/4.0/) que permite o compartilhamento do trabalho com reconhecimento da autoria e publicação inicial nesta revista.
Autores têm autorização para assumir contratos adicionais separadamente, para distribuição não exclusiva da versão do trabalho publicada nesta revista (ex.: publicar em repositório institucional ou como capítulo de livro), com reconhecimento de autoria e publicação inicial nesta revista.
Autores têm permissão e são estimulados a publicar e distribuir seu trabalho online (ex.: em repositórios institucionais ou na sua página pessoal) a qualquer momento antes ou durante o processo editorial, já que isso pode gerar alterações produtivas, bem como aumentar o impacto e a citação do trabalho publicado (disponibilizado em O Efeito do Acesso Livre no link http://opcit.eprints.org/oacitation-biblio.html).


