Seleção genética de Calophyllum brasiliense para a formação de pomares de sementes
DOI:
https://doi.org/10.20873/jbb.uft.cemaf.v4n4.schuhliPalavras-chave:
RAPD, material de propagação, MAS, guanandiResumo
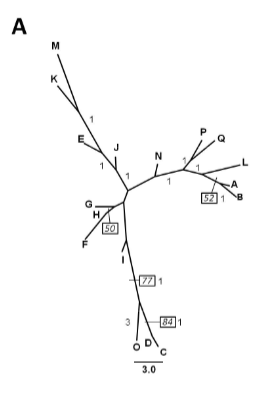
As populações de Calophyllum brasiliense encontram-se sob severa depleção e, portanto, são necessários critérios para melhorar a produção e a qualidade do material propagativo da espécie. Índices genéticos têm o potencial para orientar a redução de consangüinidade e de maximizar a representação alélica dentro das populações de interesse. Neste artigo exploramos valores genéticos para esta espécie em um pequeno relicto de floresta natural no Estado do Rio de Janeiro (Brasil). O objetivo foi o de avaliar o potencial de algumas medidas genéticas para o estabelecimento de pomares de sementes. Desde que informações genômicas de árvores nativas ainda são escassas optamos pelo uso de um marcador dominante: RAPD. O DNA de 17 árvores fenotipicamente superiores foi obtido através do método CTAB e encaminhado para amplificação por PCR. Eletroforese e documentação eletrônica foram então conduzidas. Calculamos a porcentagem de bandas polimórficas (PPB), diversidade genética (Ht), índice de informação de Shannon (i), distância genética (UPGMA) e análise de parcimônia. Seis iniciadores foram avaliados gerando 34 loci. Encontramos alta diversidade genética PPB=70,6% com Ht=0,28 e i=0,41. As relações genéticas foram apresentadas em dendrogramas (Máxima parcimônia e distância). Amostragem simulada dentro e entre agrupamentos sugerem que a amostragem dentro de grupamentos é mais eficiente para melhor capturar a diversidade genética.
Referências
Bremer K. Branch support and tree stability. Cladistics. 1994; 10: 295-304.
Caldas L. Pomares de sementes de espécies nativas – As funções das redes de sementes. In: Higa AR. and Silva LD. Pomar de sementes de espécies florestais nativas. 1st ed. FUPEF; 2006. 227-242.
Cheung WY, Hubert N, Landry BS. A simple and rapid DNA microextration method for plant, animal, and insect suitable for RAPD and other PCR analyses. Genome Res. 1993; 3: 69-70.
Cruz CD. Programa Genes: Biometria. Viçosa (MG): Editora UFV; 2006.
Kruskal JB. Multidimensional scaling by optimizing goodness of fit to a no metric hypothesis. Psychometrika. , 1964; 29(1): 1-27.
Lazar I. [homepage on the internet].Gel Analyzer. Debrecen, Hungary: University of Debrecen; updated in 2010; accessed in 2013 Jan 11. Available from: http://www.gelanalyzer.com/index.html
Manly BFJ. Randomization, bootstrap and Monte Carlo methods in Biology. London: Chapman & Hall, 1997.
Marques MCM, Joly CA. Estrutura e dinâmica de uma população de Calophyllum brasiliense Camb. em floresta higrófila do sudeste do Brasil. Rev Bras Bot. 2000; 23(1):107-12.
Muchugi A, Kadu C, Kindt R, Kipruto H, Lemurt, S, Olale K, Nyadoi P, Dawson I, Jamnadass, R. Molecular markers for tropical trees: A Practical guide to principles and procedures. ICRAF Technical Manual no. 9. World Agroforestry Centre Nairobi; 2009.
Nei M, Li W-H. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. Natl. Acad. Sci. USA. 1979; 76(10): 5269-73.
Nei M. Genetic distance between populations. Am. Nat. 1972; 106:283-92.
Newbury HJ, Ford-Lloyd BV. The use of RAPD for assessing variation in plants. Plant Growth Regul. 1993; 12:43-51, 1993.
Oliveira-Filho AT, Ratter JA. A study of the origin of central brazilian forests by the analysis of plant species distribution patterns. Edinb J Bot. 1995; 52: 141-94.
Reitz R, Klein RM, Reis A. Projeto madeira de Santa Catarina. Sellowia. 1978; 28(30): 218-24.
Rohlf FJ. NTSYS-pc v. 1.8. Numerical taxonomy and multivariate analysis system. Setauket, NY: Applied Biostatistics Inc. 1993.
Sebbenn, AM. Número de árvores matrizes e conceitos genéticos na coleta de sementes para reflorestamentos com espécies nativas. Rev Inst Flor. 2002; 14: 115-132.
Sebbenn, AM. Sistemas de reprodução em espécies arbóreas tropicais e suas implicações para a seleção de árvores matrizes para reflorestamentos ambientais. In: Higa AR. and Silva LD. Pomar de sementes de espécies florestais nativas. 1st ed. FUPEF; 2006. 93-138.
Swofford, D. L. PAUP*. Phylogenetic Analysis Using Parsimony (*and Other Methods). Version 4b10. Sunderland, Massachusetts: Sinauer Associates. 2003.
Tivang JG, Nienhuis J, Smith OS. Estimation of sampling variance of molecular marker data using the bootstrap procedure. Theor Appl Genet. 1994; 89(25):264-9.
Yeh F, Yang RC, Mao J, Ye Z, Boyle TJB. The user-friendly shareware for Population Genetic Analysis. Molecular Biology and Biotechnology Centre, University of Alberta, Edmonton, 1999.
Publicado
Como Citar
Edição
Seção
Licença
Copyright (c) 2024 - Journal of Biotechnology and Biodiversity

Este obra está licenciado com uma Licença Creative Commons Atribuição 4.0 Internacional.
Autores que publicam nesta revista concordam com os seguintes termos:
Autores mantêm os direitos autorais e concedem à revista o direito de primeira publicação, com o trabalho simultaneamente licenciado sob a Licença Creative Commons Attribution (CC BY 4.0 no link http://creativecommons.org/licenses/by/4.0/) que permite o compartilhamento do trabalho com reconhecimento da autoria e publicação inicial nesta revista.
Autores têm autorização para assumir contratos adicionais separadamente, para distribuição não exclusiva da versão do trabalho publicada nesta revista (ex.: publicar em repositório institucional ou como capítulo de livro), com reconhecimento de autoria e publicação inicial nesta revista.
Autores têm permissão e são estimulados a publicar e distribuir seu trabalho online (ex.: em repositórios institucionais ou na sua página pessoal) a qualquer momento antes ou durante o processo editorial, já que isso pode gerar alterações produtivas, bem como aumentar o impacto e a citação do trabalho publicado (disponibilizado em O Efeito do Acesso Livre no link http://opcit.eprints.org/oacitation-biblio.html).


